Muchas vías de descubrimiento de fármacos y anticuerpos se centran en proteínas de la membrana celular intrincadamente plegadas: cuando las moléculas candidatas a fármacos se unen a estas proteínas, como la llave de una cerradura, desencadenan cascadas químicas que alteran el comportamiento celular. Pero debido a que estas proteínas están incrustadas en la capa externa de las células que contiene lípidos, es difícil acceder a ellas y no pueden disolverse en soluciones a base de agua (hidrófobas), lo que dificulta su estudio.
“Queríamos sacar estas proteínas de la membrana celular, por lo que las rediseñamos como análogos solubles e hiperestables, que parecen proteínas de membrana pero son mucho más fáciles de trabajar”, explica Kasper Goverde, estudiante de doctorado en el Laboratorio de Proteínas. Diseño. e Inmunoingeniería (LPDI) en la Escuela de Ingeniería.
Brevemente, Goverde y un equipo de investigación de LPDI, dirigido por Bruno Correa, utilizaron el aprendizaje profundo para diseñar versiones sintéticas solubles de proteínas de membrana celular comúnmente utilizadas en la investigación farmacéutica. Mientras que los métodos de detección tradicionales se basan en la observación indirecta de reacciones celulares a candidatos a fármacos y anticuerpos, o en la extracción laboriosa de pequeñas cantidades de proteínas de membrana de células de mamíferos, el enfoque computacional de los investigadores les permite tratar las células mediante eliminación equivalente. Después de diseñar un análogo de proteína soluble utilizando su proceso de aprendizaje profundo, pueden usar bacterias para producir una gran cantidad de proteínas modificadas. Luego, estas proteínas pueden unirse directamente a candidatos moleculares de interés en solución.
“Nuestra hipótesis es que las proteínas solubles producen un lote de análogos. E. coli Es aproximadamente 10 veces menos costoso que utilizar células de mamíferos”, afirma el estudiante de doctorado Nicholas Goldbach.
La investigación del equipo fue publicada recientemente en la revista. La naturaleza.
Cambiando el guión del diseño de proteínas
En los últimos años, los científicos han utilizado con éxito redes de inteligencia artificial que utilizan el aprendizaje profundo para diseñar nuevas estructuras de proteínas, por ejemplo basándose en secuencias de entrada de bloques de construcción de aminoácidos, pero prediciéndolas. Pero para este estudio, los investigadores se interesaron por las capas de proteínas que ya existen en la naturaleza. Lo que necesitaban era una versión más accesible y soluble de estas proteínas.
“Tuvimos la idea de revertir el proceso de aprendizaje profundo que predice las estructuras de las proteínas: si ingresamos una estructura, ¿puede decirnos la secuencia de aminoácidos correspondiente?” Explica Goverde.
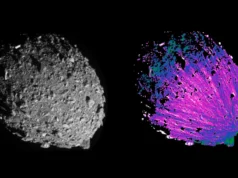
Para lograr esto, el equipo utilizó la plataforma de predicción de estructuras AlphaFold2 de Google DeepMind para generar secuencias de aminoácidos para versiones solubles de varias proteínas clave de la membrana celular en función de sus estructuras 3D. Luego, utilizaron otra red de aprendizaje profundo, Protein MpNN, para optimizar estas secuencias de proteínas solubles activas. Los investigadores se alegraron al descubrir que su enfoque demostró un éxito y una precisión notables en la producción de proteínas solubles que conservaban partes de su funcionalidad nativa, incluso cuando se aplicaban a pliegues altamente complejos que hasta ahora habían eclipsado a otros métodos de diseño.
“El Santo Grial de la Bioquímica”
Un logro notable del estudio fue el éxito de Pipeline en el diseño de un análogo soluble de una forma de proteína conocida como receptor acoplado a proteína G (GPCR), que representa alrededor del 40 por ciento de la proteína de la membrana celular humana y es un objetivo farmacéutico importante. .
“Mostramos por primera vez que podemos rediseñar la forma de un GPCR en un análogo soluble estable. Este ha sido un problema de larga data en bioquímica, porque si puedes resolverlo, tienes nuevos medicamentos que se pueden detectar muy rápidamente. y fácilmente, dice el científico del LPDI Martín Pacesa.
Los investigadores ven estos hallazgos como evidencia de la aplicación de su proyecto a la investigación de vacunas e incluso a tratamientos contra el cáncer. Por ejemplo, diseñaron un análogo soluble de un tipo de proteína llamada claudina, que desempeña un papel en hacer que los tumores sean resistentes al sistema inmunológico y a la quimioterapia. En sus experimentos, el análogo de claudina soluble del equipo conservó sus propiedades biológicas, lo que refuerza la promesa del proyecto de generar objetivos interesantes para el desarrollo farmacéutico.